この記事は次の論文の解説です。
Olaf Ronneberger, Philipp Fischer, Thomas Brox,U-Net: Convolutional Networks for Biomedical Image Segmentation,
論文の概要
この論文「U-Net: Convolutional Networks for Biomedical Image Segmentation」では、バイオメディカルイメージのセグメンテーションに特化したU-Netアーキテクチャを紹介しています。U-Netは、少数のアノテーション付きトレーニングサンプルを効果的に利用するためにデータ拡張を活用し、コンテキストを捉える収縮パスと、正確なローカライゼーションを実現する対称的な拡張パスを持つネットワークです。少数の画像からエンドツーエンドでトレーニング可能で、ISBIチャレンジにおいて従来の最良の方法を上回る成果を示しました。
1. Introduction(イントロダクション)
イントロダクションでは、畳み込みネットワークが多くの視覚認識タスクで最先端の成果を上げている背景を述べています。しかし、医療画像処理では、数千のトレーニング画像を収集するのが困難であり、各ピクセルにクラスラベルを割り当てる必要があるという課題があります。この論文は、その課題を克服するためのU-Netアーキテクチャを提案しています。
2. Related Work(関連研究)
関連研究のセクションでは、既存の手法との比較を行い、U-Netがどのようにそれらの欠点を克服するかを説明しています。Ciresanらのスライディングウィンドウ方式のネットワークは、ピクセルごとのクラスラベル予測に有効ですが、遅さと冗長性が課題です。これに対し、完全畳み込みネットワーク(FCN)を基にしたU-Netは、効率的かつ高精度なセグメンテーションを実現します。
3. Network Architecture(ネットワークアーキテクチャ)
ネットワークアーキテクチャのセクションでは、U-Netの具体的な構造を詳しく説明しています。U-Netは、収縮パスと拡張パスから成る対称的なU字型アーキテクチャで、前者はコンテキストをキャプチャし、後者は高解像度のセグメンテーションを可能にします。高解像度の特徴マップとアップサンプルされた出力を結合し、より正確なセグメンテーションを実現します。
4. Training(トレーニング)
トレーニングのセクションでは、U-Netのトレーニング方法について詳細に説明しています。ストキャスティック勾配降下法(SGD)を用い、データ拡張としてランダムな弾性変形を適用します。これにより、限られたアノテーション付き画像からでも、多様な変形に対する不変性を学習することができます。また、重み付き損失関数を用いて、接触している物体を効果的に分離します。
5. Experiments(実験)
実験のセクションでは、U-Netの性能を評価するために行った実験について報告しています。電子顕微鏡画像の神経構造セグメンテーションでは、ISBI 2012のチャレンジで従来の手法を大幅に上回る成果を達成しました。また、光学顕微鏡画像の細胞セグメンテーションでは、ISBIセル追跡チャレンジ2015で高評価を得ました。
6. Conclusion(結論)
結論のセクションでは、U-Netアーキテクチャが多種多様なバイオメディカルセグメンテーションタスクにおいて非常に優れた性能を発揮することをまとめています。少量のアノテーション付き画像でも高精度なセグメンテーションが可能であり、トレーニング時間も合理的です。U-Netは、少量のデータで高精度なセグメンテーションを実現するための有力なアプローチとして、その特性を示しています。
1. Introduction(イントロダクション)
このセクションでは、U-Netの背景と目的について詳しく説明しています。まず、畳み込みネットワーク(CNN)の近年の成功について述べています。特にKrizhevskyらの研究では、ImageNetデータセットを用いて、8層の大規模ネットワークのスーパーバイズドトレーニングが成功し、CNNのブレークスルーをもたらしました。
次に、医療画像処理における課題を取り上げています。多くの視覚タスクでは画像全体に単一のクラスラベルを割り当てますが、医療画像処理では各ピクセルにクラスラベルを割り当てる必要があります。これにより、局所的な分類が求められ、数千のトレーニング画像を収集することが困難なため、大きな障害となります。
本論文では、完全畳み込みネットワーク(FCN)を基に、少数のトレーニング画像でも効果的に動作するよう改良・拡張したU-Netを提案しています。このネットワークは、収縮パスと拡張パスから構成され、前者はコンテキストをキャプチャし、後者は高解像度のセグメンテーションを可能にします。
さらに、少ないトレーニングデータに対処するために、ランダムな弾性変形などのデータ拡張手法を強く活用し、変形に対する不変性をネットワークに学習させます。提案するU-Netは、少数の画像からエンドツーエンドでトレーニングでき、従来のスライディングウィンドウ方式のCNNを上回る性能を示します。また、GPU上で1秒未満でセグメンテーション処理が完了します。
最後に、提案するネットワークは、ISBIチャレンジで電子顕微鏡画像の神経構造セグメンテーションにおいて従来の最良の方法を上回り、光学顕微鏡画像のセグメンテーションでも優れた成果を上げました。イントロダクションは、畳み込みネットワークの成功例と医療画像処理の課題を明確にし、U-Netのアプローチとその有効性を紹介することで、論文全体の基盤となる背景情報と研究の重要性を読者に伝えています。
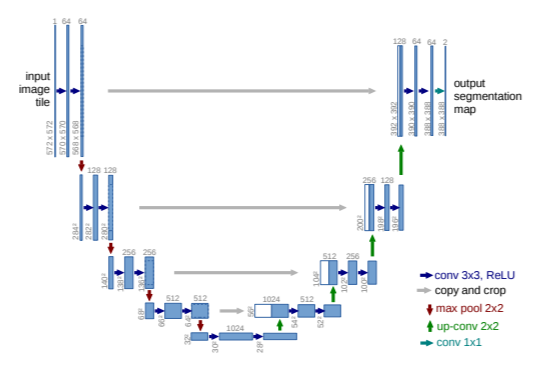
この画像は、U-Netアーキテクチャの全体像を示しています。U-Netは、画像セグメンテーションのために設計された畳み込みニューラルネットワーク(CNN)であり、主に収縮パス(Contracting Path)と拡張パス(Expansive Path)の二つの部分から構成されています。以下に各部分の詳細を説明します。
収縮パス(Contracting Path)
収縮パスは、入力画像タイルから始まり、畳み込みとプーリングを繰り返しながら特徴マップのサイズを縮小し、特徴チャネルの数を増やします。
Conv 3×3, ReLU(青い矢印): 3×3の畳み込み層を適用し、ReLU活性化関数を使用します。これにより、画像の局所的な特徴が抽出されます。
Max Pool 2×2(赤い矢印): 2×2のマックスプーリング層を適用し、特徴マップのサイズを半分に縮小します。
各段階で、特徴チャネルの数は倍増します(例: 64から128、128から256)。
拡張パス(Expansive Path)
拡張パスは、収縮パスの反対方向に進み、特徴マップのサイズを拡大し、最終的なセグメンテーションマップを生成します。
Up-conv 2×2(緑の矢印): 2×2のアップコンボリューション(逆畳み込み)層を適用し、特徴マップのサイズを倍増します。
Copy and Crop(灰色の矢印): 収縮パスから対応する特徴マップをコピーし、クロップして結合します。これにより、低レベルの詳細情報と高レベルのコンテキスト情報が組み合わされます。
Conv 3×3, ReLU(青い矢印): 再度、3×3の畳み込み層を適用し、ReLU活性化関数を使用します。
各段階で、特徴チャネルの数は半減します(例: 512から256、256から128)。
最終出力
Conv 1×1(緑の矢印): 最終層では、1×1の畳み込みを適用して各ピクセルを目的のクラス数にマッピングします。
特徴と利点
対称性: 収縮パスと拡張パスが対称的に設計されており、U字型のアーキテクチャを形成しています。これにより、ローカルな詳細情報とグローバルなコンテキスト情報が効果的に統合されます。
高解像度の特徴保持: 収縮パスからの高解像度特徴マップを拡張パスで再利用することで、精細なセグメンテーションが可能になります。
この図は、U-Netの設計と動作の概要を視覚的に示しており、その高いセグメンテーション性能を支える重要な要素を明らかにしています。
2. Related Work(関連研究)
このセクションでは、U-Netが提案された背景と、それに関連する以前の研究について述べています。U-Netの有効性を示すために、既存の手法との比較を行い、U-Netがどのようにそれらの欠点を克服するかを説明しています。
1. スライディングウィンドウ方式
Ciresanら(2012)は、スライディングウィンドウ方式を用いて各ピクセルのクラスラベルを予測するネットワークを提案しました。この方法では、各ピクセルの周囲のパッチ(局所領域)を入力として使用します。
メリット: 各ピクセルの局所的な情報をキャプチャし、セグメンテーションのためのコンテキストを提供します。
デメリット:
速度の問題: 各パッチに対して個別にネットワークを実行するため、非常に遅くなります。
冗長性: 重複するパッチが多くなるため、計算が冗長になります。
2. マルチレイヤー特徴量の利用
最近のアプローチでは、複数のレイヤーからの特徴を利用して分類を行う方法が提案されています。これにより、良好なローカライゼーションとコンテキストの使用が同時に可能となります。
例: Seyedhosseiniら(2013)やHariharanら(2014)は、階層的モデルやロジスティック・ディスジュンクティブ・ノーマル・ネットワークを使用したアプローチを提案しました。
3. 完全畳み込みネットワーク(FCN)
Longら(2014)は、完全畳み込みネットワーク(FCN)を提案しました。これは、通常の収縮ネットワークにアップサンプリングレイヤーを追加し、出力の解像度を高めるものです。
メリット: FCNは、ローカライゼーションを改善し、より正確なセグメンテーションを可能にします。
限界: しかし、FCNにはさらに改善の余地があります。
4. U-Netの改良点
本論文では、FCNのアーキテクチャを基にして、さらに改良を加えたU-Netを提案します。
シンメトリックなアーキテクチャ: U-Netは、収縮パスと拡張パスがほぼ対称的であり、U字型のアーキテクチャを持ちます。
高解像度特徴の結合: 高解像度の特徴マップとアップサンプルされた出力を結合し、より正確なセグメンテーションを実現します。
データ拡張: 特に医療画像における変形に対する不変性を学習するため、ランダムな弾性変形を用いたデータ拡張を強調しています。
結論
関連研究のセクションは、U-Netが提案された背景と、従来の方法との違いを明確にするための重要な部分です。これにより、読者はU-Netの革新性とその必要性を理解することができます。
3. Network Architecture(ネットワークアーキテクチャ)
このセクションでは、U-Netの具体的なアーキテクチャについて詳しく説明しています。U-Netは、収縮パス(Contracting Path)と拡張パス(Expansive Path)から構成される対称的なU字型のネットワークです。
収縮パス(Contracting Path)
構造: 収縮パスは、典型的な畳み込みネットワークのアーキテクチャに従い、繰り返し適用される二つの3×3畳み込み(パディングなし)、各畳み込みの後にRectified Linear Unit(ReLU)と2×2の最大プーリング(stride 2)で構成されます。
特徴マップ: 各ダウンサンプリングステップで、特徴チャネルの数は倍増します。これにより、ネットワークは入力画像のより抽象的で高次の特徴を捉えることができます。
拡張パス(Expansive Path)
構造: 拡張パスは、特徴マップのアップサンプリング(2×2の逆畳み込み)、収縮パスからの対応するクロップされた特徴マップとの結合(concatenation)、二つの3×3畳み込み(各畳み込みの後にReLU)で構成されます。
特徴マップ: アップサンプリングの各ステップで、特徴チャネルの数は半減します。これにより、高解像度のセグメンテーションを実現します。
最終レイヤー: 最終的な層では、1×1の畳み込みを使用して各64成分の特徴ベクトルを目的のクラス数にマッピングします。
全体のアーキテクチャ
対称性: U-Netのアーキテクチャは、収縮パスと拡張パスがほぼ対称的であり、これがU字型の構造を形成します。
クロップと結合: 畳み込みによる境界ピクセルの損失を補うために、収縮パスからの高解像度特徴マップをクロップして、アップサンプリングされた出力と結合します。
タイル戦略: 出力セグメンテーションマップのシームレスなタイル化を可能にするために、入力タイルサイズを慎重に選択することが重要です。これにより、大きな画像のセグメンテーションが可能となります。
特徴と利点
高解像度情報の保持: 高解像度特徴を収縮パスから拡張パスに伝播させることにより、より精細なセグメンテーションが可能です。
データ拡張: 特に医療画像では、データ拡張(例えば、弾性変形)が重要です。これにより、ネットワークは多様な変形に対する不変性を学習します。
効率性: U-Netは、GPU上で512×512ピクセルの画像を1秒未満でセグメンテーションできます。
結論
ネットワークアーキテクチャのセクションでは、U-Netの設計の詳細と、その設計がいかにして高解像度かつ効率的なセグメンテーションを可能にするかを説明しています。この対称的な収縮パスと拡張パスの構造により、U-Netは少量のトレーニングデータからでも優れたセグメンテーション性能を発揮します。
4. Training(トレーニング)
このセクションでは、U-Netのトレーニング方法について詳細に説明しています。
トレーニングデータとエネルギー関数
入力画像とセグメンテーションマップ: トレーニングには、入力画像とそれに対応するセグメンテーションマップが使用されます。これらは、Caffeのストキャスティック勾配降下法(SGD)を用いてトレーニングされます。
エネルギー関数: ピクセル単位のソフトマックスとクロスエントロピー損失関数を組み合わせたエネルギー関数が使用されます。ソフトマックス関数は各ピクセルに対するクラス確率を計算し、クロスエントロピー損失関数は予測確率と実際のラベルとの誤差を最小化します。
ピクセル単位の重み付き損失
重み付き損失: 医療画像において、接触している物体を分離することが重要です。このため、接触するセル間の背景ラベルに大きな重みを与える重み付き損失関数を使用します。
重みマップ: 各グラウンドトゥルースセグメンテーションに対して、重みマップが事前計算されます。このマップは、クラス間の頻度の違いを補正し、接触するセル間の分離境界を学習するようネットワークを強制します。
データ拡張
重要性: トレーニングサンプルが少ない場合、データ拡張が不可欠です。データ拡張により、ネットワークは必要な不変性と堅牢性を学習できます。
手法: 顕微鏡画像においては、シフトや回転不変性、変形や灰色値の変動に対する堅牢性が重要です。特に、ランダムな弾性変形は非常に効果的で、これにより、ネットワークはわずかなアノテーション付き画像からでも学習できます。
トレーニングの詳細
バッチサイズとモーメント: 入力タイルサイズを大きくし、バッチサイズを1に減らすことで、GPUメモリを最大限に活用します。高いモーメント(0.99)を使用し、過去のトレーニングサンプルの多くが現在の最適化ステップに影響を与えるようにします。
初期重みの重要性: 畳み込み層や異なるパスを持つ深層ネットワークにおいて、初期重みの良好な設定が重要です。各特徴マップの分散が単位分散に近くなるよう、ガウス分布から初期重みを引きます。
結論
トレーニングのセクションでは、U-Netのトレーニング方法、損失関数、データ拡張の重要性、初期重み設定の重要性について詳細に説明しています。これにより、U-Netが少量のトレーニングデータからでも高精度なセグメンテーションを実現できる理由を明確にしています。
5. Experiments(実験)
このセクションでは、U-Netの性能を評価するために行った実験について詳細に説明しています。具体的には、電子顕微鏡(EM)画像と光学顕微鏡(LM)画像を用いたセグメンテーションタスクについて報告しています。
1. EM画像の神経構造セグメンテーション
データセット: ISBI 2012のEMセグメンテーションチャレンジで提供された、ドロソフィラ(ショウジョウバエ)の幼虫神経構造の連続切片電子顕微鏡画像30枚(512×512ピクセル)を使用しました。各画像には、セル(白)と膜(黒)の完全なアノテーションが含まれています。
評価方法: セグメンテーションマップを閾値10レベルで評価し、「ワーピングエラー」、「ランドエラー」、「ピクセルエラー」を計算します。
結果: U-Netは、7つの回転バージョンの入力データを平均して使用し、ワーピングエラー0.0003529とランドエラー0.0382を達成しました。これは、Ciresanらのスライディングウィンドウ方式のネットワークを上回る結果です。
2. 光学顕微鏡画像の細胞セグメンテーション
データセット: ISBI 2014および2015のセル追跡チャレンジで提供された2つのデータセットを使用しました。
PhC-U373: グリオブラストーマ・アストロサイトーマU373細胞を相差顕微鏡で記録した35枚の部分的にアノテーションされた画像。
DIC-HeLa: DIC顕微鏡で記録した平滑なガラス上のHeLa細胞の20枚の部分的にアノテーションされた画像。
評価方法: IOU(Intersection over Union)を用いてセグメンテーションの精度を評価しました。
結果:
PhC-U373: U-Netは平均IOU 92%を達成し、次点のアルゴリズム(83%)を大きく上回りました。
DIC-HeLa: U-Netは平均IOU 77.5%を達成し、次点のアルゴリズム(46%)を大幅に上回りました。
実験結果の図示
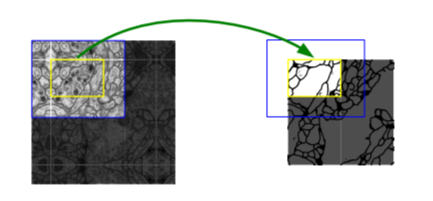
出典: Olaf Ronneberger, Philipp Fischer, Thomas Brox,U-Net: Convolutional Networks for Biomedical Image Segmentation,
図2(Overlap-tile strategy): 任意の大きさの画像のシームレスなセグメンテーションのためのオーバーラップタイル戦略を示しています。これは、入力画像の欠けている部分をミラーリングによって補間することにより、大きな画像に対しても適用可能です。
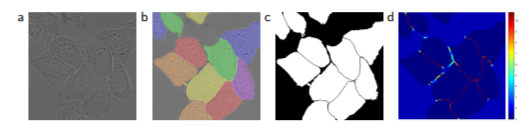
出典: Olaf Ronneberger, Philipp Fischer, Thomas Brox,U-Net: Convolutional Networks for Biomedical Image Segmentation,
図3(HeLa cells on glass): DIC顕微鏡で記録したHeLa細胞の生画像、グラウンドトゥルースセグメンテーション、生成されたセグメンテーションマスク、ピクセル単位の損失重みマップを示しています。
結論
実験のセクションでは、U-Netが非常に異なるバイオメディカルセグメンテーションタスクにおいて優れた性能を発揮することを示しています。特に、少量のアノテーション付きトレーニングデータから高精度なセグメンテーションを実現し、従来の方法を大幅に上回る結果を得ています。
6. Conclusion(結論)
結論のセクションでは、U-Netアーキテクチャの全体的な成果とその意義についてまとめています。
U-Netのパフォーマンス
U-Netは、非常に異なるバイオメディカルセグメンテーションタスクにおいて優れたパフォーマンスを示しました。具体的には、電子顕微鏡画像の神経構造セグメンテーションおよび光学顕微鏡画像の細胞セグメンテーションで、既存の方法を大幅に上回る成果を上げました。
データ拡張とトレーニング効率
データ拡張の重要性: ランダムな弾性変形を用いたデータ拡張により、少量のアノテーション付きトレーニングデータからでもネットワークが高精度なセグメンテーションを学習できることが確認されました。これは、特に医療画像における変形に対する不変性を学習するために重要です。
効率的なトレーニング時間: NVidia Titan GPU(6GB)を用いた場合、トレーニング時間は約10時間と非常に合理的であることが示されました。
実装と適用性
実装の提供: 論文で提案されたU-Netの完全なCaffeベースの実装およびトレーニング済みネットワークは公開されており、他の研究者が容易に利用できるようになっています。
多くのタスクへの適用可能性: U-Netアーキテクチャは、バイオメディカルセグメンテーション以外の多くのセグメンテーションタスクにも容易に適用できることが期待されます。
将来の展望
さらなる応用: 提案されたアーキテクチャとトレーニング手法は、他の様々なセグメンテーションタスクにも応用できる可能性があります。今後の研究において、U-Netの適用範囲がさらに広がることが期待されます。
結論の要点
結論のセクションでは、U-Netアーキテクチャの強み、特に少量のデータで高精度なセグメンテーションが可能である点、トレーニングの効率性、および多くのタスクへの適用可能性を強調しています。また、実装が公開されているため、他の研究者が容易にこのアプローチを試すことができる点も重要です。これにより、U-Netが今後の研究や実践において広く利用されることを期待しています。
まとめ
本論文「U-Net: Convolutional Networks for Biomedical Image Segmentation」では、医療画像処理のために特化されたU-Netアーキテクチャを提案しています。このネットワークは、少数のアノテーション付きデータを効率的に利用するために、データ拡張技術を活用し、収縮パスと拡張パスから構成される対称的なU字型構造を持ちます。U-Netは、電子顕微鏡画像や光学顕微鏡画像を用いたセグメンテーションタスクにおいて、既存の手法を大幅に上回る成果を示し、特に少量のトレーニングデータからでも高精度なセグメンテーションを実現することが確認されました。また、論文で提案された実装は公開されており、他の研究者が容易に利用できる点も強調されています。これにより、U-Netは多様なバイオメディカルセグメンテーションタスクに広く適用され、今後の研究や実践において重要な役割を果たすことが期待されます。
参考文献
Olaf Ronneberger, Philipp Fischer, Thomas Brox,U-Net: Convolutional Networks for Biomedical Image Segmentation,
